Cut&Tag-seq
分析項目 | 類別 | 分析 | 備注 |
原始數(shù)據(jù)整理、過濾及質(zhì)量評估 | A | √ |
|
與參考基因組序列比對 | A | √ |
|
Peak Calling | A | √ |
|
基因附近及Peak中心信號分布圖 | A | √ |
|
Peak鄰近基因功能分析 | A | √ |
|
差異Peak序列Motif及注釋分析 | A | √ |
|
差異Peak鄰近基因功能分析 | A | √ |
|
Overlap基因功能分析 | A | √ |
|
Peak Calling
使用統(tǒng)計學(xué)方法計算出參考基因組上比對上的 Reads 顯著富集的區(qū)域(稱為 Peak),這些區(qū)域在不同的實驗中表示不同的研究重點,獲得 Peak 后,可對 Peak 進行后續(xù)的 Peak 序列 Motif 分析、 Peak 注釋等分析,進一步挖掘感興趣的方向。Peak 在外顯子、內(nèi)含子、5’UTR、3’UTR、基因間區(qū)等區(qū)域上的分布情況如下:
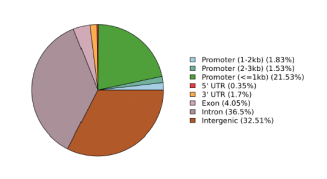
功能性區(qū)域Peak分布
Peak序列Motif分析
轉(zhuǎn)錄因子往往傾向于結(jié)合在特定的 DNA 序列上,這些 DNA 序列通常具有高度相似的核苷酸序列模式,即每個轉(zhuǎn)錄因子都有一個目標 DNA 序列的 Motif,公開發(fā)表的轉(zhuǎn)錄因子數(shù)據(jù)庫中一般記錄了轉(zhuǎn)錄因子對應(yīng)的 Motif 信息。對 Peak 序列計算 Motif,在公共數(shù)據(jù)庫中搜索這些 Motif 對應(yīng)的轉(zhuǎn)錄因子,即可得到此種狀態(tài)下染色質(zhì)開放區(qū)可能結(jié)合的轉(zhuǎn)錄因子。

Motif分析示意圖
Peak鄰近基因GO富集分析
統(tǒng)計Peak鄰近基因顯著富集的GO條目,從而展示可能被轉(zhuǎn)錄因子調(diào)控的基因功能,對每個Peak鄰近基因進行GO注釋,得到其所有的GO功能條目。
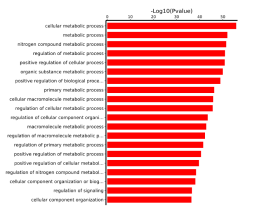
GO富集柱狀圖
差異Peak鄰近基因Pathway富集分析
統(tǒng)計各個KEGG Pathway上包含的差異Peak鄰近基因數(shù)目及富集程度,進而確定差異Peak鄰近基因主要參與的代謝途徑和信號通路。
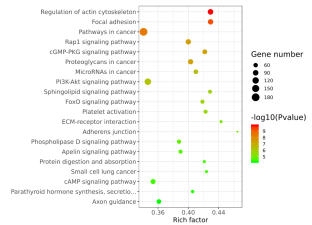
KEGG富集分析
Overlap基因分析
為了研究轉(zhuǎn)錄因子/組蛋白修飾是如何調(diào)控下游基因,通常要結(jié)合表達數(shù)據(jù) RNA-seq 分析,進一步研究轉(zhuǎn)錄因子/組蛋白修飾的調(diào)控作用,即促進或抑制基因表達。
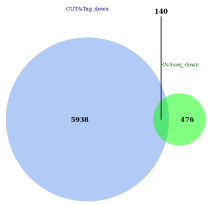
韋恩圖

























